Immer mehr Antibiotika-resistente Bakterienstämme stellen die Medizin vor eine große Herausforderung. Denn bei vielen Infektionen, die eigentlich mal einfach zu behandeln waren, versagen die gängigen Antibiotika. Neue antibakterielle Arzneimittel werden so dringend benötigt wie selten zuvor, aber dennoch läuft die Entwicklung neuer Antibiotika sehr langsam. Wieso das so ist, und welche Strategien vielleicht trotzdem zum Erfolg führen könnten, lest ihr hier.
Enges vs. breites Spektrum
Wie ich in Teil 1 beschrieben habe, ist die Entstehung von immer mehr Antibiotika-resistenten Bakterienstämmen im Prinzip unvermeidbar, solange wir Antibiotika verwenden; wenn ihr wissen wollt wieso, dann lest Teil 1 doch gerne zuerst.
Die Tatsache, dass die Zahl der Resistenzen zunimmt, bedeutet natürlich auch, dass wir neue Behandlungsoptionen brauchen. Wie die Aussehen könnten, und welche Probleme es dabei gibt, beschreiben zum Beispiel zwei spannende Reviews von Cook und Wright und Bergkessel et al. Neue Antibiotika sind aber gar nicht so einfach zu finden: Antibiotika müssen oft in hohen Dosen eingesetzt werden und dürfen daher auf keinen Fall zu toxisch sein. Außerdem müssen sie anderen physikochemischen Eigenschaften entsprechen als „normale“ Arzneistoffe, weil sie ja nicht nur von Menschen aufgenommen werden und in ihnen wirken sollen, sondern auch zusätzlich in die Bakterien gelangen und dort wirken müssen. Vor allem gibt es aber sehr viele unterschiedliche Bakterien und je breiter das Wirkspektrum sein soll – also gegen wie viele verschiedene Bakterien ein Stoff wirkt – desto schwieriger wird es.
Breitband-Antibiotika haben einen entscheidenden Vorteil: Sie brauchen viel weniger Diagnostik. Während vor dem Einsatz von Stoffen mit engem Spektrum erst die Erreger identifiziert werden müssen, können Breitband-Antibiotika ohne diesen Nachweis verwendet werden. Und auch wenn nicht unnötigerweise breit therapiert werden sollte, fehlt für einen Erregernachweis oft die Zeit (häufig beruht der Nachweis noch auf der Anzucht der Bakterien, was einige Tage dauern kann). Allerdings sind neue Breitband-Antibiotika viel schwieriger zu finden als solche mit engem Spektrum. Daher könnte die Zukunft der antibiotischen Therapie aus mehr Stoffen mit engem Spektrum in Kombination mit schnelleren (point of care) Nachweisen der Erreger bestehen.
Neue Antibiotika aus der Natur
Woher sollen die neuen Antibiotika kommen? – Und ich meine damit wirklich neue Antibiotika, mit neuen Zielstrukturen und Wirkmechanismen, statt Derivaten bereits bekannter Stoffe. Eine Möglichkeit wäre der Ort, von dem auch viele der etablierten Antibiotika kommen: Aus der Natur. Denn tatsächlich produzieren viele Mikroorganismen antibakterielle Stoffe und die meisten Antibiotika sind entweder direkt mikrobiellen Ursprungs oder davon abgeleitet.
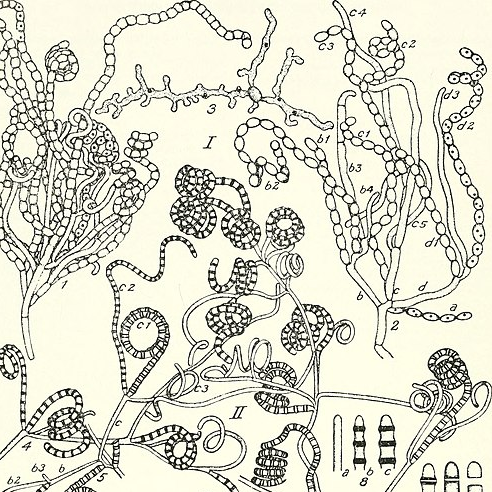
Bei den Antibiotikaproduzenten stechen vor allem die Actinomyceten hervor (also um genau zu sein Bakterien der Ordnung der Actinomycetales, aber ehrlicherweise verwirrt mich Systematik von Bakterien immer etwas). Um dann aber neue Stoffe zu finden und nicht nur bekannte, müssen neue Techniken verwendet werden. Methoden wie die Genomik bieten gute Ansatzpunkte jenseits der phänotypischen Testung, also im Prinzip einfach des „Ausprobierens“, ob die Anwesenheit bestimmter Bakterien oder Stoffe das Wachstum anderer Bakterien hemmt. So kann zum Beispiel nach potentiellen Biosynthese-Genclustern neuer antibakterieller Stoffe gesucht werden oder nach bestimmten Resistenzgenen – die meisten Antibiotika-produzierenden Mikroorganismen tragen nämlich auch Resistenzen dagegen in ihrem Erbgut. Es könnte sich aber auch lohnen, abseits der Actinomyceten zu suchen und andere Mikroorganismen unter die Lupe zu nehmen, die bisher nicht so ausführlich nach neuen Antibiotika untersucht wurden.
So viele Möglichkeiten die genomischen Methoden auch bieten, sollte man dabei die besonderen Anforderungen an Antibiotika nicht aus den Augen verlieren. Tausend neu identifizierte Biosynthese-Gene bringen nichts, wenn die entsprechenden Stoffe die Zellwand von Bakterien nicht überwinden können oder von den Bakterien direkt wieder aus der Zelle herausgeschleust werden. Zusätzlich spielt auch der Kontext der Infektion eine Rolle: Bakterien verhalten sich bei einer Infektion unter Umständen anders als in einer Laborkultur und dementsprechend kann auch der Effekt eines potentiellen Antibiotikums unterschiedlich sein.
Alte Arzneistoffe als neue Antibiotika
Naturstoffe sind aber natürlich nicht die einzige Möglichkeit, um neue Antibiotika zu finden. Auch komplett synthetische Stoffe können Potential haben, wie die sogenannten Oxazolidinone zeigen, eine Gruppe synthetischer Antibiotika, zu denen u.a. das klinisch sehr wichtige Linezolid gehört.
Neue (synthetische) Antibiotika-Kandidaten müssen allerdings nicht unbedingt neue Moleküle sein. Es gibt ein Methode namens drug repurposing, bei der existierende Arzneistoffe für neue Einsatzgebiete verwendet werden. Ein erfolgreiches Beispiel ist Zidovudin, ein Wirkstoff bei HIV-Infektionen, das ursprünglich jedoch gegen Krebs entwickelt wurde. Tatsächlich sind etwa 30 % der zugelassenen Arzneistoffe der letzten Jahre in solcher Weise wiederverwendet – allerdings kein einziges Antibiotikum.
Viele existierende Wirkstoffe zeigen zumindest in vitro antibakterielle Eigenschaften, auch wenn das für ihre in vivo-Anwendung an Menschen noch nicht viel heißen muss. Der Vorteil des drug repuposings ist jetzt, dass für diese Stoffe häufig schon Daten zur Toxizität, zur Pharmakokinetik, der Maximaldosis usw. bekannt sind. Dadurch ist es viel einfacher abzuschätzen, ob sie sich tatsächlich auch als klinisch nützliche Antibiotika eigenen würden, und falls ja ist der Weg dahin sowohl günstiger als auch schneller.
Und selbst falls die antibakterielle Wirkung allein zu gering sein sollte, könnten solche Stoffe eventuell als Adjuvans eingesetzt werden, die in Kombination die antibakterielle Wirkung eines anderen Wirkstoffs verstärken oder Resistenzen umgehen, ohne selbst (stark) antibakteriell zu sein.
Bakteriophagen – Viren gegen Bakterien
Einige Menschen werden sich jetzt wohl Fragen: Wann geht es endlich um Phagen? Bakteriophagen sind eine Art Trendthema, wenn es um Alternativen zu Antibiotika geht. Es sind Viren, die ausschließlich Bakterien befallen und damit prinzipiell perfekt um selektiv Bakterien abzutöten. Genau darauf haben sie sich schließlich über Millionen Jahre an Evolution entwickelt.
Phagen infizieren immer nur spezifische Arten von Bakterien, wodurch sie z.B. gegenüber des Mikrobioms sehr schonend sind. Diese Selektivität ist aber nicht nur ein Vorteil: Phagen haben das gleiche Problem wie Antibiotika mit engem Spektrum. Sie brauchen einen ausführlichen Erregernachweis, was nicht immer praktikabel ist. Außerdem sind Phagen potentiell immunogen, können also vom Immunsystem erkannt werden und Abwehrreaktionen wie Fieber auslösen.
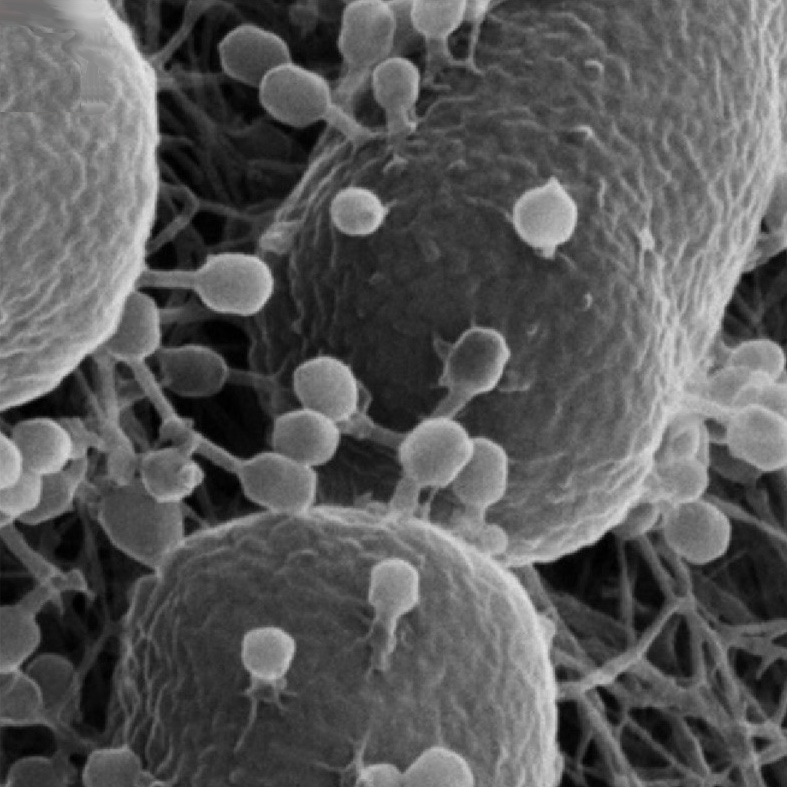
Ein Mythos ist, dass Phagen keine Probleme mit der Entwicklung von Resistenzen hätten. Zwar haben sie sich sehr lange entwickelt, um Bakterien zu infizieren und zu töten, aber die Bakterien hatten genauso lange Zeit, um Abwehrmechanismen zu entwickeln. Kombinationen verschiedener Phagen könnten Resistenzen verzögern, und weiterentwickelte Varianten könnten eine bestehende Resistenz überwinden, aber im Prinzip haben Phagen, was die Resistenzentwicklung angeht, nicht wirklich Vorteile gegenüber Antibiotika.
Das soll allerdings nicht heißen, dass die Phagentherapie keinen Platz in der Behandlung von Infektionen hätte. Gerade wenn sich auch die Identifikation von Erregern weiterentwickelt, können Phagen eine wichtige Rolle bei Infektionen mit Antibiotika-resistenten Bakterien spielen. Dass der Einsatz von Phagen eine hoch-personalisierte Therapie und damit aufwändig und teuer ist, und daher auch Antibiotika nicht ersetzen kann, wird sich jedoch nicht so schnell ändern.
Das wirtschaftliche Problem
Das größte Hindernis bei der Entwicklung neuer Antibiotika habe ich jedoch noch gar nicht angesprochen. Es ist allerdings kein wissenschaftliches Problem, sondern ein wirtschaftliches.
Mit neuen Antibiotika verdient nämlich niemand Geld. Die Entwicklung von Arzneimitteln ist – vor allem durch klinische Studien – extrem teuer. Für den Entwicklungsprozess, von der Entdeckung einer Leitstruktur bis zur Einführung in die Klinik, muss ein Unternehmen etwa 1,5 Milliarden Euro investieren, im Verkauf sind Antibiotika aber eher Pfennigware. Außerdem sollten neue Antibiotika im Idealfall so selten wie möglich eingesetzt werden; sie sollten als Reserve-Arzneimittel für dringende Fälle vorbehalten werden, damit sich nicht direkt neue Resistenzen dagegen entwickeln. Das alles führt nur leider dazu, dass Unternehmen mit neuen Antibiotika ihre Investitionskosten nicht wieder reinholen können, und daher auch keinen Anreiz dazu haben, Antibiotika zu entwickeln.
Tatsächlich wird daher überproportional viel Arbeit in diesem Bereich von der akademischen Forschung übernommen, viel mehr als bei anderen Arzneimittelgruppen. Allerdings haben die Universitäten bei weitem nicht genug Mittel, diese Aufgabe allein zu übernehmen.
Wir haben in unserer Gesellschaft entschieden, dass neue Arzneimittel von gewinnorientierten Unternehmen entwickelt werden und diese Forschung nicht von der öffentlichen Hand finanziert wird. Dieses Modell funktioniert jetzt – leider im Angesicht von mehr und mehr Resistenzen – allerdings nicht mehr.
Eine dreiteilige Lösung
Wir brauchen also eine dreiteilige Lösung für das Resistenz-Problem: Der erste Teil ist eine wissenschaftliche Lösung für die Entwicklung neuer antibakterieller Wirkstoffe. Dazu gehört ein besseres Verständnis für die physikochemischen Eigenschaften von Antibiotika, neue Quellen für Leitstrukturen wie z.B. bisher wenig untersuchte Mikroorganismen oder das drug repurposing, und sinnvolle Plattformen zur Evaluation der antibakteriellen Wirkung, die verschiedene bakterielle Physiologien und Infektionskontexte nicht außer Acht lassen.
Der zweite Teil ist eine therapeutische Lösung. Sie muss eine schnellere und einfachere Identifikation von Erregern, den gezielten Einsatz von Antibiotika mit engem Spektrum und personalisierten Therapien, die Verwendung von Adjuvantien und Hemmstoffen von Resistenzmechanismen, und ein sinnvolles Therapiemanagement zur Verlangsamung der der Resistenzentstehung umfassen. Außerdem ist eine der besten Methoden gegen Antibiotika-resistente Erreger die Vermeidung von Infektionen. Das ist natürlich selbst ein großes Feld, aber klar ist, dass Impfungen auch im Rennen gegen Antibiotika-Resistenzen extrem wichtig sind.
Der dritte Teil ist schließlich eine wirtschaftliche und politische Lösung. Neue Antibiotika kosten Geld, und dieses Geld müssen wir irgendwie aufbringen. Wir können nicht erwarten, dass unser auf Gewinn ausgelegtes Gesundheitssystem Therapien hervorbringt, die mehr kosten als sie einbringen. Und diese wirtschaftliche Lösung muss immer auch global gedacht werden: Auch in ärmeren Ländern muss die Versorgung mit (neuen) antibakteriellen Arzneimitteln sichergestellt sein. Resistenzen können überall auf der Welt entstehen und Bakterien halten sich nicht an Staatsgrenzen, und deshalb sind neue Antibiotika nur für reiche Industrienationen niemals ausreichend.
Wenn euch dieser Beitrag gefallen hat, abonniert doch gerne meinen Newsletter, um keine Blogposts mehr zu verpassen. Und hier geht es nochmal zum ersten Teil dieses Textes über die Entstehung von Resistenzen und Resistenzmechanismen, falls ihr ihn noch nicht gelesen habt.
Pingback:Wieso Antibiotika-Resistenzen unvermeidbar sind – Antibiotika-Resistenzen, Teil 1 - PharmBlog